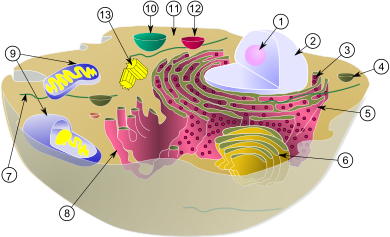
Diagramme d'une
cellule animale typique, les mitochondries sont indiquées par la légende 9
L'ensemble des mitochondries d'une cellule constitue ce que l'on appelle son
chondriome.
En
1857,
Kölliker décrit les aspects de la mitochondrie dans le
muscle. En
1890,
Altmann décrit une technique de coloration des mitochondries qu'il appelle
bioblastes et postule leur autonomie métabolique et
génétique. En
1937, un scientifique allemand,
Hans Adolf Krebs, construit un modèle qu’il appela «
citric acid cycle ». Ce cycle a lieu dans la mitochondrie chez les
eucaryotes. En
1940-
43, Claude isole les mitochondries dans des cellules du
foie. En
1948-
50, Kennedy et Lajdurkhfekzh
Lehninger montrent que le
cycle de Krebs, la
bêta-oxydation et la
phosphorylation oxydative ont tous lieu dans la mitochondrie. En
1978,
Peter Mitchell obtient le
Prix Nobel pour sa
théorie chimiosmotique. En
1981,
Anderson et son équipe découvrent la structure génétique de l’
ADN mitochondrial humain. Finalement,
Boyer et
Walker, eux aussi, obtiennent le Prix Nobel pour leurs études sur la structure et le fonctionnement de l'
ATP synthétase.

Schéma descriptif de la structure mitochondriale :
1 : membrane interne.
2 : membrane externe.
3 : espace inter-membranaire.
4 : matrice.
Les mitochondries ont une dimension de 1-2 à
10 μm de long et de 0,5 à
1 μm de diamètre. Elles se composent de 2 membranes mitochondriales, une externe et une interne, qui délimitent trois milieux : le milieu extra-mitochondrial (
cytoplasme de la
cellule), l'espace inter-membranaire et la matrice. Chacune est de l'ordre des
6 nm et l'espace intermembranaire est de
7 nm.
- La membrane externe est formée de 70 % de protéines et de 30 % de lipides polaires. Elle contient de nombreuses protéines appelées porines (VDAC) qui forment des canaux aqueux au travers de la membrane. La porine (protéine transmembranaire composée de 16 feuillets béta formant les canaux protéiques traversant la couche bimoléculaire de lipides) laisse passer toutes les molécules hydrophiles d'une masse moléculaire inférieure à 10 000 daltons1 (anions, cations, les acides gras, le pyruvate, les nucléotides le traversent). La membrane externe présente des complexes TOM constitués de plusieurs sous-unités protéiques dont des récepteurs et des canaux aqueux qui permettent l'entrée des protéines d'origine nucléaire dans la mitochondrie ou l'insertion de ces mêmes protéines dans la membrane externe.
- La membrane interne est beaucoup moins perméable que la membrane externe. Elle est composée de 75 % de protéines et de 25 % de lipides. Elle contient en quantité un phospholipide double, la cardiolipine, renfermant 4 acides gras rendant cette membrane imperméable aux ions. Les autres molécules doivent passer par un transporteur pour traverser la membrane interne. La membrane interne présente des complexes TIM 23, TIM 22, et OXA. Le TIM 23 permet l'entrée de protéines situées dans l'espace inter-membranaire dans la matrice mitochondriale et dans la membrane interne. Le TIM 22 permet l'insertion des protéines dans la membrane interne et notamment des protéines à plusieurs domaines transmembranaires. Le complexe OXA permet la sortie de la matrice pour certaines protéines d'origine mitochondriale.
La membrane interne forme des invaginations qui apparaissent sous forme de crêtes ou replis au microscope électronique. Ces crêtes augmentent la surface de la membrane et donc la capacité de phosphorylation oxydative. Grâce à cette caractéristique on peut déduire que si une mitochondrie possède beaucoup de crêtes c'est que la cellule a besoin d'une grande quantité d'énergie et donc elle pourra produire plus d'ATP (cellule en activité). On retrouve également à son niveau des protéines de transport spécifiques pour les petites molécules utilisées par la matrice, les enzymes de la chaîne respiratoire, l'ATP-synthase ou complexe F0-F1 visible au microscope électronique sous forme de protubérance interne.
Une mitochondrie ne peut provenir que de la croissance et de la division d'une autre mitochondrie déjà existante. Normalement, avant la division cellulaire, la mitochondrie double sa masse puis se scinde en deux. Elles sont aussi capables de fusionner entre elles. Cette division débute par l'apparition d'un sillon de division sur la membrane interne. Elle a lieu pendant toute l'interphase et nécessite l'intervention de la protéine DRP1 (voisine de la
dynamine). La
réplication de l'ADN mitochondrial n'est pas limitée à la phase S du
cycle cellulaire. Le nombre de mitochondries par cellule est régulé par l'activité cellulaire. Par exemple, une cellule musculaire au repos contient 5 à 10 fois moins de mitochondries qu'une
cellule musculaire activée en permanence.
Le fait que la mitochondrie possède son ADN propre, comme les
chloroplastes, indique une origine exogène : il est maintenant admis que les mitochondries proviennent de l'endosymbiose d'une α-protéobactérie il y a environ 2 milliards d'années. La théorie
endosymbiotique de l'origine des mitochondries, a été développée et argumentée par
Lynn Margulis dès
1966, puis a été appuyée par la découverte de l'ADN spécifique des mitochondries en
1980. Il semble qu'au cours de l'évolution l'ADN originel de la bactérie ait subi diverses évolutions, perdu un grand nombres de gènes, parfois transféré dans l'ADN de la cellule hôte. Parallèlement à ce report de la synthèse de certaines protéines vers l'hôte, ce dernier a développé un arsenal de translocases, enzymes permettant le transfert de ces protéines vers la matrice mitochondriale.
Génome mitochondrial[modifier]

Vue détaillée d'une mitochondrie
Selon la théorie endosymbiotique, les mitochondries possèderaient une origine monophylétique unique. Une cellule eucaryote primitive (ou une
Archaea) aurait intégré un
endosymbiote procaryote il y a environ 1,5 à 2 milliards d’années, lorsque l’atmosphère primitive s’est enrichie en oxygène
2,3. Les études phylogénétiques indiquent que cet endosymbionte est apparenté aux alpha-protéobactéries, le plus proche parent de la mitochondrie connu actuellement étant
Rickettsia prowazekii, un parasite intracellulaire obligatoire
2. Au cours de l’évolution, la majorité des gènes de l’endosymbionte originel auraient été perdus ou bien transférés vers le noyau de la cellule eucaryote hôte
3,4. En effet, les nombreux
pseudogènesmitochondriaux présents dans le génome attestent d’un processus de transfert tout au long de l’évolution
5,6.
Le matériel génétique (
ADN mitochondrial) de la mitochondrie (qui est la seule partie des cellules animales à posséder son propre ADN, en plus du noyau) sert souvent dans les recherches
phylogénétiques. Le génome mitochondrial (ADNmt) humain est circulaire et composé de 16 569 paires de bases, dont 13
cistrons codant des
ARNm, 22 gènes pour des
ARNt et 2 gènes pour des
ARNr.
Le génome mitochondrial peut être très différent d'une espèce à l'autre, il est extrêmement dynamique, il est souvent hétéroplasmique, c'est-à-dire que différentes formes coexistent au sein de la même cellule. Il peut être trouvé sous forme circulaire ou linéaire, double ou simple brin. Ces différentes formes sont, entre autres, les produits de la réplication du génome mitochondrial par un mécanisme de cercle roulant, mais aussi d'un mécanisme de réplication recombinaison-dépendant, similaire à la réplication du phage T4. Les génomes mitochondriaux sont habituellement représentés sous forme circulaire, le « cercle maître » qui correspond à la molécule décrivant le mieux le génome.
Les ribosomes mitochondriaux ou mitoribosomes sont différents des ribosomes de la cellule : ils sont plus petits (70S au lieu de 80S).
Le code génétique employé pour la synthèse des protéines peut être différent de celui utilisé dans les synthèses
cytosoliques. Chez les vértébrés 4
codons sur 64 ont une signification différente, dont le codon UGA qui est transcrit dans le cytosol en codon stop mais dans la matrice UGA est transcrit en tryptophane (Trp/W), AGG et AGA codent un codon STOP au lieu d'une arginine (Arg/R) et AUA code la méthionine (Met/M) au lieu de l'isoleucine (Ile/I). L'ADN mitochondrial peut aussi se répliquer.
Chez les animaux, lors de la reproduction sexuée, les mitochondries du spermatozoïde pourraient passer dans l'
ovocyte, mais le nombre de mitochondries ainsi transférées reste très faible en comparaison de celles déjà présentes dans l'ovocyte. Autrement dit, la quasi totalité des mitochondries de la cellule-œuf provient du gamète femelle. L'étude de l'
ADN mitochondrial humain permet donc de retracer les relations généalogiques entre les individus seulement selon la voie maternelle. Certaines études ont ainsi pu décrire un
génome mitochondrial ancestral duquel descendraient tous les génomes mitochondriaux de l'humanité. L'individu femelle supposé qui portait ce
génome a été dénommé
Ève mitochondriale. Ce terme biblique reste toutefois trompeur, il est en effet très peu probable que l'humanité ait un unique ancêtre féminin et de récentes études, prouvant le transfert de mitochondries provenant des spermatozoïdes lors de la fécondation, remettent en cause cette théorie.
Le protéome mitochondrial est l'ensemble des protéines présentes dans les mitochondries d'une cellule eucaryote à un moment donné. Le protéome est un ensemble dynamique défini dans le temps (moment considéré : stade de développement, matin ou soir) et dans l'espace (échantillon considéré : cellule, tissu, organisme). Pour décrire l'ensemble des protéines pouvant être présentes dans une mitochondrie à un moment quelconque de la vie de l'organisme, on utilisera le terme de protéome total.
Le protéome mitochondrial est composé de protéines produites dans les mitochondries et codées dans le génome mitochondrial, et de protéines produites dans le cytoplasme et codées dans le génome nucléaire. La plupart des complexes
enzymatiques (exemple : ATP-synthase) sont formés par la juxtaposition de
polypeptides synthétisés dans la mitochondrie et dans le
cytosol (le fluide interne de la cellule).
Bien que le les mitochondries soient les descendantes de bactéries, les protéines de leur protéome ne sont pas toutes d'origine bactérienne, Ainsi chez la
levure 50 à 60 % des protéines mitochondriales ont des homologues chez les
procaryotes alors que 40 à 50 % n’en ont pas
3.
Protéines mitochondriales codées par le génome mitochondrial[modifier]
Suivant les organismes 1 à 10 % des protéines mitochondriales sont directement synthétisée dans la matrice par les mitoribosomes, à partir de l'
ADN mitochondrial.
Protéines mitochondriales codées par le génome nucléaire[modifier]
Les protéines mitochondriales possédant un homologue
procaryote résultent probablement du transfert des
gènes de l’
endosymbionte vers le
nucléaire tandis que les
protéines non homologues à des protéines procaryotes résultent d’un phénomène « d’enrichissement » du
protéome mitochondrial par de nouvelles protéines et donc de nouvelles fonctions
2.
Les protéines mitochondriales codées par le génome nucléaire (ou protéines mitochondriales nucléaires) sont importées à l'intérieur de la matrice mitochondriale par différents mécanismes possibles :
- des complexes d'importation (3 sur la membrane interne, 2 sur la membrane externe);
- un peptide signal (environ 15 à 30 acides aminés) en position N-terminale de la protéine qui permet sa reconnaissance et son importation dans la mitochondrie7,8 ;
- grâce à un apport énergétique.
La taille du
protéome mitochondrial humain est estimée à plus d’un millier de protéines, dont environ 1% codées par le génome mitochondrial (13 protéines)
9, dont actuellement la moitié est identifiée
10,11. Seules 13 protéines sont codées par l’
ADN mitochondrial, vestige du génome de l’
endosymbionte. Toutes les autres
protéines sont codées par le
génome nucléaire.
Elle est considérée comme la « centrale énergétique » de la cellule, car c'est là que se déroulent les dernières étapes du
cycle respiratoire qui convertit l'énergie des molécules organiques issues de la digestion (
glucose) en énergie directement utilisable par la
cellule (
ATP). En cas d'absence d'oxygène la cellule utilise la
fermentation dans le cytoplasme pour produire l'énergie nécessaire à son fonctionnement, mais c'est un système beaucoup moins efficace, qui dégrade de façon incomplète le substrat. La production d'acide lactique donne lieu, par exemple, à des phénomènes de crampes. L'augmentation de la concentration en ions H+ dans les cellules musculaires est une des raisons de la fatigue après une activité intense. En effet, ces ions H+ changent le pH intracellulaire et modifient de fait les conditions de fonctionnement enzymatiques de la cellule qui ne peut plus travailler correctement.
C'est dans la mitochondrie que se déroulent les 2 dernières phases de la
respiration cellulaire : le
cycle de Krebs (dans la matrice) et la
chaîne de transport d'électrons (au niveau de la membrane interne). La première étape, la
glycolyse, se déroule dans le cytoplasme cellulaire. Via le
cycle de Krebs (donc en condition d'aérobiose), la mitochondrie permet, à partir d'une molécule de glucose, la production de 36 ou 38 molécules d'
ATP(cela dépend de la navette utilisée pour transporter le NAD de la glycolyse).
Les mitochondries participent à l'
apoptose (mort cellulaire) avec le cytochrome C. De plus, elles ont aussi une fonction de concentration et de stockage des ions calcium, sodium et potassium où ils sont stockés sous forme de granules opaques. On trouve également de l'or, du fer et de l'osmium.
Poisons mitochondriaux[modifier]
Certains poisons ont pour rôle non pas d'empêcher les différents complexes de fonctionner, c'est-à-dire que les transferts d'électron de la chaîne respiratoire sont effectués mais ces protéines, les découplants ou UCP vont court-circuiter le complexe V (ATP synthase) en créant un canal à travers la membrane interne. Ce pore permet aux protons de passer de l'espace inter-membranaire vers la matrice dans le sens de leur gradient, ce qui se traduit par un dégagement de chaleur mais aucune production d'ATP. Exemple : Dinitrophénol
Maladies mitochondriales[modifier]
Notes et références[modifier]
- ↑ Pierre Cau, Raymond Seïte et Andrée Robaglia-Schlupp, Cours de Biologie cellulaire, Ellipses, 1999
- ↑ a, b et c (en) S.G. Andersson, A. Zomorodipour, J.O. Andersson, T. Sicheritz-Ponten, U.C. Alsmark, R.M. Podowski, A.K. Naslund, A.S. Eriksson, H.H. Winkler & C.G. Kurland, « The genome sequence of Rickettsia prowazekii and the origin of mitochondria », dans Nature, vol. 396, no 6707, novembre 1998, p. 133–140 [lien PMID [archive], lien DOI [archive]]
- ↑ a, b et c (en) M.W. Gray, G. Burger & B.F. Lang, « The origin and early evolution of mitochondria », dansGenome Biology, vol. 2, no 6, 5 juin 2001, p. reviews1018.1–1018.5 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) C.G. Kurland & S.G. Andersson, « Origin and evolution of the mitochondrial proteome », dansMicrobiology and Molecular Biology Reviews, vol. 64, no 4, décembre 2000, p. 786–820 [lien PMID [archive]]
- ↑ (en) Y. Tourmen, O. Baris, P. Dessen, C. Jacques, Y. Malthiery, & P. Reynier, « Structure and chromosomal distribution of human mitochondrial pseudogenes », dans Genomics, vol. 80, no 1, juillet 2002, p. 71–77 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) Woischnik, M. and C.T. Moraes, « Pattern of organization of human mitochondrial pseudogenes in the nuclear genome », dans Genome Research, vol. 12, no 6, juin 2002, p. 885–893 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) Rusch, S.L. & D.A. Kendall, « Protein transport via amino-terminal targeting sequences: common themes in diverse systems », dans Molecular Membrane Biology, vol. 12, no 4, octobre 1995, p. 295–307 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) G. Schatz & B. Dobberstein, « Common principles of protein translocation across membranes », dansScience, vol. 271, no 5255, mars 1996, p. 1519–1526 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) M.F. Lopez, B.S. Kristal, E. Chernokalskaya, A. Lazarev, A.I. Shestopalov, A. Bogdanova, & M. Robinson, « High-throughput profiling of the mitochondrial proteome using affinity fractionation and automation », dansElectrophoresis, vol. 21, no 16, octobre 2000, p. 3427–3440 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) C. Andreoli, H. Prokisch, K. Hortnagel, J.C. Mueller, M. Munsterkotter, C. Scharfe, & T. Meitinger, « MitoP2, an integrated database on mitochondrial proteins in yeast and man », dans Nucleic Acids Research,vol. 32, no Database issue, janvier 2004, p. D459–D462 [lien PMID [archive], lien DOI [archive]]
- ↑ (en) D. Cotter, P. Guda, E. Fahy, & S. Subramaniam, « MitoProteome: mitochondrial protein sequence database and annotation system », dans Nucleic Acids Research, vol. 32, no Database issue, janvier 2004,p. D463–D467 [lien PMID [archive], lien DOI [archive]]