Un virus est une entité biologique nécessitant un hôte, souvent une cellule, dont il utilise les constituants pour se multiplier. On le considère de plus en plus comme faisant partie des acaryotes.
Les virus existent sous une forme extracellulaire (unité matérielle indépendante appelée alors virion) ou intracellulaire (virus intégré sous forme dormante ou détournant activement la machinerie cellulaire au profit de sa réplication). Sous la forme intracellulaire (à l'intérieur de la cellule hôte), les virus sont des éléments génétiques qui peuvent se répliquer de façon indépendante par rapport au chromosome, mais non indépendamment de la cellule hôte. Sous la forme extracellulaire, les virus sont des objets particulaires, infectieux, constitués au minimum d'un acide nucléique englobé dans une capside de protéines.
Le débat sur la nature des virus (vivants ou pas) repose sur des notions complexes et reste aujourd'hui ouvert. Cependant, selon certaines définitions courantes du vivant (entité matérielle réalisant les fonctions de relation, nutrition, reproduction), les virus ne seraient donc pas des êtres vivants.[réf. nécessaire]
La virologie est la science qui étudie les virus. Elle est étudiée par des virologues ou virologistes.
Le mot virus est issu du latin virus, qui signifie « poison ». Se terminant par un s, il ne prend pas de marque particulière au pluriel en français1.
Sommaire[masquer] |
Découverte[modifier]
Les maladies virales comme la rage, la fièvre jaune, la variole, affectent les humains depuis des siècles. Des hiéroglyphes mettent en évidence la poliomyélite dans l'Égypte antique, les écrits de l’Antiquitégréco-romaine et d’Extrême-Orient décrivent certaines maladies virales.
À la fin du xixe siècle, la conception d’agents infectieux qui n’étaient ni des bactéries, ni des champignons, ni des parasites, invisibles au microscope optique, était encore difficile.
À cette époque, les scientifiques isolaient des agents infectieux à travers des filtres de porcelaine utilisés pour recueillir les bactéries. Entre 1887 et 1892, le botaniste russe Dimitri Ivanovski étudia une maladie végétale, la mosaïque du tabac, et montra que la sève des plantes malades contenait un agent infectieux qui n’était pas retenu par les filtres Chamberland conçus par le biologiste du même nom. Ivanovski pensait qu’il s’agissait d’une toxine ou bien d’une très petite bactérie. C’est le chimiste hollandais Martinus Beijerinck qui approfondit ces travaux et, en 1898, écarta non seulement l’hypothèse bactérienne mais aussi l'hypothèse toxinique : diluant la sève de plantes infectées il l'inocula à des plantes qui développèrent la maladie; réitérant la manipulation il put transmettre la maladie de multiples fois et démontrer que la sève de la dernière plante infectée était aussi virulente que la première, effet qu'une toxine, après tant de dilutions n'aurait pu produire2. Beijerinck appela l'agent Contagium vivum fluidum ('soluble living germ'). À la même époque, le virus de la fièvre aphteuse est le premier virus identifié par Friedrich Löffler et Paul Frosch. Le virus de la fièvre jaune est le premier virus pathogène de l’Homme identifié entre 1900 et1902. Pasteur les nomma « infrabactéries », d'autres les qualifiaient de « virus filtrants » ou « virus ultrafiltrants ».
C’est pendant la Première Guerre mondiale que l’anglais Frederick Twort et le microbiologiste franco-canadien Félix d'Hérelle mettent en évidence le phénomène de « lyse transmissible » observable par la lyse des bactéries cultivées en milieu solide. Ce phénomène est dû à un virus de bactéries que Félix d'Hérelle baptisa bactériophage. Les virus des plantes, des animaux, de l’Homme et des bactéries étaient ainsi découverts et leurs listes ne cessèrent de s’allonger au cours du xxe siècle.
Vers 1925, la définition de virus3 devint « un agent responsable d'une maladie infectieuse, parasite, de nature particulaire et de taille comprise entre 0.01 et 0.3 microns »4.
L’apparition de la microscopie électronique dans les années 1930 permit l’observation des virus, mais on ne savait toujours pas à cette époque ce qu’ils étaient réellement. Le biochimiste américain Wendell Stanley cristallisa le virus de la mosaïque du tabac sous forme de cristal protéique en 1935. L'année suivante, des études complémentaires montrèrent que ce cristal contenait également de l’ARN. Les études ultérieures montrèrent que selon les virus étudiés, ceux-ci étaient composés soit de protéines et d’ARN, soit de protéines et d’ADN. C’est en 1957 qu'André Lwoff proposa une définition claire et moderne des virus.
À partir des années 1960, le développement des cultures cellulaires, de la microscopie électronique, puis de la biologie moléculaire permit aux scientifiques de progresser dans la compréhension des mécanismes de réplication des virus, dans la réalisation de diagnostics fiables et dans l’élaboration de vaccins.
Caractéristiques[modifier]
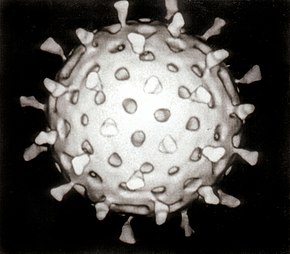
Un virus se caractérise par son incapacité à se multiplier par division. Il a besoin pour cela d'infecter une cellule hôte pour utiliser sa machinerie : un virus est unparasite intracellulaire obligatoire. Il est composé d'une ou plusieurs molécules d'acide nucléique (soit d'ADN, soit d'ARN, simple ou double brin), entourées d'une coque de protéines appelée capside, parfois d'une enveloppe. Il ne possède en général aucune enzyme pouvant produire de l'énergie. Les virus étaient historiquement considérés comme des particules organiques de petite taille (inférieure à celle d'une bactérie), en règle générale moins de 250 nanomètres. Toutefois de nouveaux virus récemment découverts, comme mimivirus avec sa taille de 400 nm et la possession à la fois d'ADN et d'ARN, remettent en cause cette vision historique.
La forme libre du virus (ou particule virale) s'appelle le virion.
Nature[modifier]
Il y a débat sur la nature des virus.
Les virus possèdent des constituants en commun avec les cellules vivantes, comme un acide nucléique (ADN ou ARN) et des protéines. Cependant, selon la définition du biochimiste Wendell Stanley, les virus sont de « simples » associations de molécules biologiques. Ils sont le fruit d’une auto-organisation de molécules organiques et ne sont donc pas vivants. François Jacob insiste aussi sur cette caractéristique des virus : « placés en suspension dans un milieu de culture, ils ne peuvent nimétaboliser, ni produire ou utiliser de l’énergie, ni croître, ni se multiplier, toutes fonctions communes aux êtres vivants5 ». Les virus ne peuvent se multiplier qu’en utilisant l’équipement enzymatique d’une cellule vivante. De plus, les virus contiennent bien un acide nucléique, de l’ADN ou de l’ARN, mais pas les deux (sauf le mimivirus, évoqué plus haut), à la différence des cellules vivantes.
Au cours des dernières années, des entités intermédiaires sont découvertes : le mimivirus, infectant une amibe, possède dans son génome 1 200 gènes (davantage que certaines bactéries). Certains de ces gènes participeraient à la synthèse protéique et à des mécanismes de réparation de l’ADN6. Il existe chez le mimivirus une trentaine de gènes présents habituellement chez les organismes cellulaires mais absents chez les virus. Le virus ATV d’archées présente lui aussi des caractéristiques étonnantes : ce virus en forme de citron présente la particularité de se modifier en dehors du contexte cellulaire par un mécanisme actif. Il est capable de s’allonger à chaque extrémité à une température de 80 °C, température à laquelle vit son hôte Acidianus à proximité des sources hydrothermales7. Néanmoins organes et échanges cycliques, donc métabolisme, restent absents.
Les virus ont aussi un rôle dans l’évolution. Patrick Forterre avance même l’hypothèse que les virus seraient les « inventeurs » de l’ADN8. À l’origine de la vie, l’ARN dominait (hypothèse du monde à ARN) et assurait à la fois les fonctions de stockage et transmission de l’information génétique et de catalyse des réactions chimiques. L’ADN serait apparu ensuite et sélectionné en raison de sa plus grande stabilité. D’après Patrick Forterre, le premier organisme à ADN serait un virus. L'ADN conférerait au virus le pouvoir de résister à des enzymes dégradant les génomes à ARN, arme de défense probable des protocellules. On retrouve le même principe chez des virus actuels, qui altèrent leur ADN pour résister à des enzymes produites par des bactéries infectées.
Le débat sur le caractère vivant ou inerte des virus reste encore aujourd’hui ouvert.[réf. nécessaire] Répondre à cette question exige de répondre au préalable à une autre : qu’est-ce que la vie ? D’après Ali Saïb,« la notion du vivant est une notion dynamique, évoluant en fonction de nos connaissances. En conséquence, la frontière entre la matière inerte et le vivant est tout aussi instable »9. L'existence ou non d'un métabolisme, c'est-à-dire d'un ensemble cohérent de processus chimiques l'homéostasie (et non la reproduction) constitue un discriminant possible, en tout cas commode.
Structure[modifier]
Une particule virale complète, appelé virion, est composée d’un filament d’acide nucléique, généralement stabilisé par des nucléoprotéines basiques, enfermé dans une coque protéique protectrice appeléecapside. La forme de la capside est à la base des différentes morphologies des virus. La taille des virus se situe entre 10 et 400 nm. Les génomes des virus ne comportent que de quelques gènes à 1 200 gènes. Le plus petit virus connu est le virus delta, qui parasite lui-même celui de l'hépatite B. Il ne comporte qu'un seul gène. Le plus gros virus connu est le mimivirus, avec un diamètre qui atteint 400 nanomètres et un génome qui comporte 1 200 gènes.
Le virus a une forme microscopique variable, si la représentation "usuelle" lui donne l'image du V.I.H., les différentes espèces sont de formes allants de la sphère à l'apparence insectoïde.
Acide nucléique[modifier]
Le filament d'acide nucléique peut être de l'ADN ou de l'ARN. Il représente le génome viral. Il peut être circulaire ou linéaire, bicaténaire (double brin) ou monocaténaire (simple brin). Le génome sous forme d'ADN est généralement bicaténaire. Le génome sous forme d'ARN est généralement monocaténaire et peut être à polarité positive (dans le même sens qu'un ARN messager) ou à polarité négative (complémentaire d'un ARN messager). Le peloton central d'acide nucléique est dénommé nucléoïde.
Capside[modifier]
La capside est une coque qui entoure et protège l'acide nucléique viral. La capside est constituée par l'assemblage de sous-unités protéiques appelées capsomères. L'ensemble de la capside et du génome est nommé nucléocapside. La structure de la capside peut présenter plusieurs formes. On distingue en général deux groupes principaux de virus : les virus à symétrie cubique (ou à capside icosaédrique) et les virus à symétrie hélicoïdale.
Enveloppe[modifier]
De nombreux virus sont entourés d'une enveloppe (ou péplos) qui prend naissance au cours de la traversée des membranes cellulaires. Sa constitution est complexe et présente un mélange d'éléments cellulaires et d'éléments d'origine virale. On y trouve des protéines, des glucides et des lipides. Les virus possédant une enveloppe sont les virus enveloppés. Les virus ne possédant pas d'enveloppe sont lesvirus nus.
| Virus icosaédriques | |
| La capside icosaédrique entraîne une apparence sphérique du virus. Les protomères sont organisés en capsomères, disposés de manières régulières et géométriques. Une capsomère est composée de cinq ou six protomères : appelées pentons aux sommets et hexons au niveau des faces et des arrêtes.
Parmi les virus icosaédriques, les parvovirus ont une capside formée de 12 capsomères, les poliovirus 32 capsomères, les papillomavirus 72 capsomères tandis que la capside des adénovirus est constituée de 252 capsomères.
| |
| Virus hélicoïdaux | |
| Ces virus sont de longs cylindres (300 à 400 nm), creux, composés d’un type de protomère enroulé en spirale hélicoïdale formant des anneaux appelés capsomères. Ils peuvent être rigide ou flexible. Le matériel génétique est logé à l’intérieur du tube. Le virus de la mosaïque du tabac est un exemple de virus hélicoïdal très étudié. | |
| Virus enveloppés | |
| En plus de la capside, certains virus sont capables de s’entourer d’une structure membranaire empruntée à la cellule hôte. Cette enveloppe membranaire est composée d’une bicouche lipidique qui peut posséder des protéines codées par le génome viral ou le génome de l’hôte. Cette enveloppe donne quelques avantages aux virions par rapport à ceux composés d’une capside seule, comme la protection vis-à-vis d’enzymes ou de composés chimiques. Les virus enveloppés sont par contre plus fragiles dans l'environnement extérieur, sensibles aux détergents et à la dessiccation. Les glycoprotéines, formant des spicules, fonctionnent comme des récepteurs permettant de se fixer sur des cellules hôtes spécifiques.
Le virus de la grippe (famille des Orthomyxoviridae), le VIH, virus du SIDA (famille des Retroviridae), sont des exemples de virus enveloppés.
| |
| Virus complexes | |
| Ces virus possèdent une capside symétrique qui n’est ni hélicoïdale, ni vraiment icosaédrique. Les bactériophages comme le phage T4 d’Escherichia coli sont des virus complexes possédant une tête icosaédrique liée à une queue hélicoïdale à laquelle sont attachés des poils et des fibres caudales.
Le poxvirus (variole, vaccine) est aussi un exemple de virus complexe. C'est le virus animal parmi les plus grands (250 à 350 nm de long sur 200 à 250 nm de large). Certains virus se présentent sous formes bacillaires. C'est le cas du virus de la rage (famille des Rhabdoviridae) et du virus Ebola.
|
Réplication[modifier]
Multiplication virale[modifier]
Les virus ne peuvent se répliquer qu’au sein de cellules vivantes. C’est l’interaction du génome viral et de la cellule hôte qui aboutit à la production de nouvelles particules virales. L’infection d’une cellule par un virus, puis la multiplication du virus peuvent se résumer en différentes étapes. Toutefois, après pénétration du virus dans la cellule, ces étapes peuvent différer selon la nature du virus en question et notamment selon qu’il s’agit d’un virus à ADN ou d’un virus à ARN.
- Adsorption du virus au contact de la membrane de la cellule infectée, grâce à des récepteurs spécifiques
- Pénétration dans la cellule
- Décapsidation (libération de l'acide nucléique)
- Réplication du génome viral
- Synthèse de protéines virales
- Assemblage et encapsidation des particules virales produites
- Libération des virions hors de la cellule-hôte
Culture des virus[modifier]
Afin de mieux connaître leur biologie, leur multiplication, leur cycle de reproduction et éventuellement afin de préparer des vaccins, il est nécessaire de cultiver les virus. Ceux-ci peuvent se multiplier uniquement au sein de cellules vivantes. Les virus infectant les cellules eucaryotes sont cultivées sur des cultures de cellules obtenues à partir de tissus animaux ou végétaux. Les cellules sont cultivées dans un récipient en verre ou en plastique, puis sont infectées par le virus étudié. Les virus animaux peuvent aussi être cultivés sur œufs embryonnés et parfois chez l’animal, lorsque la culture in vitro est impossible. Les virus bactériens peuvent également être cultivés par inoculation d’une culture bactérienne sensible. Les virus de végétaux peuvent aussi être cultivés sur des monocouches de tissus végétaux, des suspensions cellulaires ou sur des plantes entières.
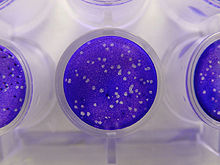
Les virus peuvent ensuite être quantifiés de différentes manières. Ils peuvent être comptés directement grâce à la microscopie électronique. Dans le cas des virus bactériens, la technique des plaques (ou plages) est très utilisée pour évaluer le nombre de virus dans une suspension. Une dilution de suspension virale est ajouté à une suspension bactérienne, puis l’ensemble est réparti dans des boîtes de Petri. Après culture, des zones claires (plages) à la surface de la gélose sont la conséquence de la destruction d’une bactérie et des bactéries adjacentes par un virion.
Les virus peuvent être purifiés grâce à diverses méthodes de biochimie (centrifugation différentielle, précipitation, dénaturation, digestion enzymatique).
Origine[modifier]
Tout être vivant peut être infecté par un virus. Il existe des virus de bactéries (les bactériophages), des virus d'Archaea, des virus d'algues (Phycodnaviridae), des virus de plantes, des virus fongiques, des virus d'animaux, parmi lesquels on trouve de nombreux agents pathogènes, et même des virus de virus10.
Il existe plusieurs hypothèses concernant l'origine et l'évolution des virus. Il est probable que tous les virus ne dérivent pas d'un ancêtre commun et les différents virus peuvent avoir des origines différentes.
- Les virus et les cellules ont pu apparaître dans la soupe primordiale en même temps et évoluer parallèlement. Dans ce scénario, au début de l’apparition de la vie, les plus anciens systèmes génétiques d'auto-réplication (probablement de l'ARN) sont devenus plus complexes et se sont enveloppés dans un sac lipidique pour aboutir au progénote à l'origine des cellules. Une autre forme réplicative aurait pu garder sa simplicité pour former des particules virales.
- Les virus pourraient dériver de cellules ayant subi une régression. D'après cette hypothèse, les ancêtres des virus auraient été des êtres vivants libres ou des micro-organismes devenus des prédateurs ou des parasites dépendants de leur hôte. Les relations de parasitisme entraînent la perte de nombreux gènes (notamment les gènes pour le métabolisme apportés par l'hôte). Cet organisme aurait co-évolué avec la cellule hôte et n'aurait conservé que sa capacité à répliquer son acide nucléique et le mécanisme de transfert de cellule à cellule. Cette hypothèse s'appuie notamment sur l'existence des rickettsies, petites bactéries ayant régressé à un tel point qu'elles ne peuvent survivre que dans une cellule hôte, et rappelant les virus.
- Les virus peuvent avoir pour origine des morceaux d'acides nucléiques qui se sont « échappés » du génome cellulaire pour devenir indépendants. Ce phénomène pourrait avoir eu lieu lors d’erreurs au cours de la réplication du matériel génétique. Les virus pourraient aussi avoir pour origine des plasmides (molécules d’ADN circulaires) ou des transposons (séquences d'ADN capables de se déplacer et de se multiplier dans un génome).
Rôle dans l'évolution[modifier]
Les virus ont un rôle naturel important de véhicule pour le transfert de gènes entre des espèces différentes, ce qui accroit la diversité génétique, et permet de disséminer des innovations génétiques au-delà de la descendance de l'individu porteur de cette mutation génétique11. On pense que les virus ont joué un rôle central dans les premiers temps de l'évolution, avant la diversification entre bactéries, archaea et eucaryotes et à l'époque du Dernier ancêtre commun universel de la vie sur terre. Les virus sont encore l'un des plus grands réservoirs sur la Terre de diversité génétique inexplorée12.
Virus et maladies[modifier]
Les virus possèdent différents mécanismes leur octroyant diverses possibilités stratégiques d'infection, dont l'incidence provoque éventuellement des maladies. Le virion pénètre une cellule hôte plus ou moins spécifique où il se désagrège, libérant son contenu qui en s'activant prend le pas sur les fonctions cellulaires normales. A ce niveau, les effets cytopathogènes des virus peuvent entraîner divers effets néfastes. Les capacités de synthèse protéique de la cellule infectée peuvent être détournées ou inhibées, tandis que la chromatine est fragmentée par des enzymes virales. Des particules virales s’accumulent dans lecytoplasme avant de s'assembler en virions. La surcharge virale endo-cellulaire provoque enfin la mort de la cellule hôte par lyse, libérant les virions qui vont ensuite disséminer.
Lorsque le virus pénètre dans une cellule non permissive, il ne peut pas se multiplier. Son génome peut cependant subsister sous la forme d’un épisome libre ou intégré au génome cellulaire. Il y a transformation cellulaire virale lorsque le génome du virus entre en interaction avec l’ADN du génome cellulaire. On appelle ces virus des virus oncogènes. Parmi ceux-ci, les rétrovirus, en s’intégrant dans le génome cellulaire, peuvent devenir tumorigène et éventuellement entraîner des cancers.
La capacité d’un virus d’entraîner une maladie est décrite en termes de pouvoir pathogène tandis que son intensité est exprimée en termes de virulence. La classification des principaux groupes de virus, et leurs correspondances en pathologie, se trouvent dans l'encyclopédie médicale Vulgaris. Cette classification est notamment basée sur le type de molécule d'acides nucléiques (ARN ou ADN) dont est constituée le virion.[3]
Maladies humaines[modifier]
Le rhume, la grippe, la varicelle, la rougeole, la mononucléose infectieuse sont des exemples de pathologies humaines relativement courantes d'origine virale. On connaît d'autres exemples plus nocifs comme le SIDA, le SRAS, la grippe aviaire, la variole, ou le virus Ebola qui entraîne des fièvres hémorragiques.
Quelques exemples de virus pathogènes pour Homo sapiens :
- VIH, virus du SIDA
- Rétrovirus
- Coxackie A virus
- Ebola
- Virus de la variole
- Virus de la grippe
- Virus de la fièvre jaune
- Virus du Nil occidental
- Cytomégalovirus
- Rotavirus
- Virus de l'Hépatite C
- Virus simien 40 ou SV40
Prévention et traitements[modifier]
Étant donné que les virus utilisent la machinerie cellulaire de l’hôte pour se reproduire à l’intérieur même de la cellule, il est difficile de les éliminer sans tuer la cellule hôte. Des médicaments antiviraux permettent cependant de perturber la réplication du virus.
Une autre approche est la vaccination qui permet de résister à l’infection.
Divers médicaments permettent de traiter les symptômes liés à l’infection. Les patients demandent souvent[réf. nécessaire] à leurs médecins qu’ils leurs prescrivent desantibiotiques, mais ils sont sans effet sur les virus. Les antibiotiques interfèrent en effet avec des constituants ou le métabolisme des bactéries et permettent donc de traiter seulement les maladies d’origine bactérienne et non les maladies d'origine virale.
Diverses méthodes de désinfection in vitro permettent d’inactiver les virus (hypochlorite de sodium à 1 %, éthanol à 70 %, glutaraldéhyde à 2 %, formaldéhyde).
Biotechnologie[modifier]
Les virus présentant en général un matériel génétique simpliste, ce sont d'excellents outils dans l’étude de la biologie moléculaire et la biologie cellulaire. Ils permettent la manipulation de fonctions cellulaires, ce qui permet d'en approfondir notre compréhension et d'éluder certains mécanismes moléculaires de la génétique comme laréplication de l'ADN, la transcription, les modifications post-transcriptionnelles de l’ARN, la traduction, le transport des protéines et l’immunologie.
Les virus peuvent être utilisés comme vecteur de gène au sein de cellules cibles. Outil utilisé par exemple pour faire acquérir à une cellule la capacité de produire une protéine d'intérêt ou pour étudier l’effet de l’introduction du nouveau gène dans le génome.
Certains virus sont utilisés en thérapie génique pour soigner diverses maladies d'origine génétique. Il s'agit par exemple de remplacer un gène défectueux provoquant des troubles fonctionnels ou mécaniques.
Les virus sont également utilisés dans la lutte contre le cancer. Certains virus sont capables de détruire spécifiquement des cellules cancéreuses.[réf. nécessaire]
Classification[modifier]
Les virus sont classifiés selon la nature de l'acide nucléique de leur génome (ADN ou ARN), la structure de l'acide nucléique (monocaténaire ou bicaténaire), la forme de l'acide nucléique (linéaire, circulaire, segmenté ou non). Les données morphologiques peuvent également être prises en compte (présence ou absence d'enveloppe, symétrie de la capside). Souvent, le sérogroupage est encore utilisé pour raffiner la définition des différences entre virus très proches.
Virus de procaryotes[modifier]
Il existe deux catégories de virus de procaryotes selon le type d’hôte qu’ils parasitent. La première catégorie regroupe ceux qui infectent les bactéries et sont appelés bactériophages. La deuxième catégorie regroupe ceux qui infectent les Archaea. Il existe quatre grands groupes morphologiques de virus de procaryotes.
- Les virus à symétrie binaire. Ce groupe représente près de 96 % des virus de procaryotes et correspond aux familles des Myoviridae, des Siphoviridae et des Podoviridae.
- Les virus à symétrie cubique avec une capside icosaédrique mais pas de queue comme les Microviridae.
- Les virus à symétrie hélicoïdale qui ont une forme de filaments comme les Inoviridae comme le phage M13.
- Les virus pléomorphes, sans capsides véritable mais possédant une enveloppe. Ce groupe rassemble six familles de virus dont cinq regroupent des virus infectant seulement les archées. Certains virus d’archées sont pléomorphes, alors que d’autres ont des formes de bouteilles, de citron, de fuseau13.
Les bactériophages possèdent un rôle dans les écosystèmes. Par exemple, dans les écosystèmes aquatiques, ils participent au contrôle de l’abondance et de la diversité bactérienne14.
Virus d'eucaryotes[modifier]
Virus d'animaux[modifier]
En principe spécifique d'une espèce ou d'un groupe de phylums génétiquement proches, les virus ont tendance à infecter un type cellulaire ou tissulaire principal ou exclusif. Cependant, il existe de nombreux virus, comme la rage, qui sont moins spécifique à un hôte par comparaison avec d'autres virus comme la maladie de Carré, le virus de l’immunodéficience féline ou la variole. Les virions se propagent principalement par contact direct entre individus, mais peuvent aussi diffuser sous forme aérosol ou par les excrétions diverses (vomis, urines, selles, larmes...) et d'éventuels parasites (moustiques, tiques, puces...).
Virus des plantes[modifier]
La structure des virus des plantes ou phytovirus, est similaire à celle des virus bactériens et animaux. Beaucoup de virus végétaux se présentent sous la forme de minces et longues hélices. La majorité ont un génome composé d’ARN. Les virus de végétaux peuvent être disséminés par le vent ou par des vecteurs comme lesinsectes et les nématodes, parfois par les graines et le pollen. Les virus peuvent aussi contaminer la plante par l’intermédiaire d’une blessure ou d’une greffe. Différents types de symptômes peuvent apparaître sur la plante infectée. Les virus peuvent provoquer des taches ou des flétrissements sur les feuilles et les fleurs. Des tumeurs peuvent survenir sur les tiges ou les feuilles.
Le virus de la mosaïque du tabac (TMV ou tobamovirus) est un exemple très étudié de virus de végétaux.
Le virus de la mosaïque du tabac (TMV ou tobamovirus) est un exemple très étudié de virus de végétaux.
Virus des insectes[modifier]
Les baculovirus sont des virus d’insectes très étudiés. Ils infectent principalement les lépidoptères. La larve de l’insecte s’infecte en ingérant de la nourriture. À partir du tube digestif, l’infection peut se transmettre aux autres tissus. L'utilisation de virus pathogènes d'invertébrés dans la lutte contre les insectes ravageurs des cultures et des forêts pourraient être l'un des moyens pour limiter ou remplacer les insecticides chimiques.
Les baculovirus sont aussi utilisés en biologie moléculaire pour exprimer un gène étranger (protéine recombinante) dans des cultures de cellules d'insecte.
Par ailleurs, certains virus de végétaux sont transmis par des invertébrés mais ne se multiplient pas chez ces vecteurs.
Les baculovirus sont aussi utilisés en biologie moléculaire pour exprimer un gène étranger (protéine recombinante) dans des cultures de cellules d'insecte.
Par ailleurs, certains virus de végétaux sont transmis par des invertébrés mais ne se multiplient pas chez ces vecteurs.
Virus des mycètes[modifier]
Les virus des champignons sont particuliers car ils se propagent lors de la fusion cellulaire. Il n'y a pas de virions extracellulaires. Chez les levures comme Saccharomyces, les virus sont transmis au moment du brassage cytoplasmique lors de la fusion cellulaire. Les champignons filamenteux comme Penicillium ou le champignon de Paris Agaricus bisporus peuvent également être infectés par des virus.ce qui peut entraîner des problèmes lors de production. Il a été imaginé d'utiliser ces virus dans le cadre d'une lutte biologique contre des champignons pathogènes.
Virus de virus[modifier]
Récemment découvert (2008)15, Sputnik 16 est un cas à part capable d'infecter un autre virus (Mamavirus) appartenant à la classe des virus géants 17 (génome de plus de 300 000 pb et taille supérieure à 0,2 μm).
On connait aussi d'autres virophages comme Mavirus18 associé à CroV 19 (un virus géant infectant Cafeteria roenbergensis).
Notes et références[modifier]
- Le pluriel latin de virus n'est pas connu. Bien que se terminant par "-us", on le considère comme un nom neutre de la 2e déclinaison ; son pluriel serait alors "vira". La forme "viri" est une faute grammaticale : c'est le pluriel des mots masculins de la 2e déclinaison ; "virii" est un barbarisme. En anglais, la marque du pluriel pour un mot se terminant par "s" est "-es" ; la forme "viruses" se retrouve d'ailleurs le plus souvent dans la littérature médicale et professionnelle.
- Page 111 du livre Une histoire des microbes [archive] de Patrick Berche, 2007, ISBN 978-2-7420-0674-8
- La première mention du terme virus apparaît chez Virgile avec le sens de "liquide sanieux et purulent"[1] [archive]
- http://hal.archives-ouvertes.fr/docs/00/04/59/36/PDF/tel-00004194.pdf [archive]
- François Jacob, Qu’est-ce que la vie ? in La Vie, Université de tous les savoirs, Editions Odile Jacob, 2002.
- Raoult D., Audic S., Robert C., Abergel C., Renesto P., Ogata H., La Scola B., Suzan M., Claverie J.-M., The 1.2-megabase genome sequence of Mimivirus, Science, 2004 Nov 19;306(5700):1344-50.
- M. Haring et al., Independant virus development outside a host, Nature, vol. 436, p. 1101-1102, 2005
- Patrick Forterre, Three RNA cells for ribosomal lineages and three DNA viruses to replicate their genomes : a hypothesis for the origin of cellular domain, PNAS, vol. 103 (10), pp. 3669-3674, 2006
- Ali Saïb, Les virus, inertes ou vivants ? in Pour la Science, décembre 2006.
- découverte d'un virophage [archive]
- Canchaya C, Fournous G, Chibani-Chennoufi S, Dillmann ML, Brüssow H ; Phage as agents of lateral gene transfer, Curr. Opin. Microbiol.,volume 6, issue 4, pages 417–24 (Août 2003) ,pmid 12941415, doi 10.1016/S1369-5274(03)00086-9
- Forterre P, Philippe H;"The last universal common ancestor (LUCA), simple or complex?", The Biological Bulletin, volume 196, issue 3, pages 373–5, 375–7, Juin 1999, pmid 11536914, doi 10.2307/1542973,[2] [archive]
- Prangishvili, D., P. Forterre, and R. A. Garrett. 2006. Viruses of the Archaea: a unifying view. Nat Rev Microbiol 4:837-48.
- K. E. Wommack and R. R. Colwell (2000) Virioplankton: Viruses in Aquatic Ecosystems. Microbiol. Mol. Biol. Rev. 64, 69-114
- Virology: A Virus gets a Virus [archive], Nature Reviews, 2008
- Siyang Sun, « Structural Studies of the Sputnik Virophage », dans Journal of Virology, Journal of Virology,vol. 84, no 2, janvier 2010, p. 894–897 [lien PMID [archive], lien DOI [archive]]
- Correspondance, Nature Reviews Microbiology, vol.7, pp. 615-625, 2009
- Fischer MG, Matthias G. and Curtis A. Suttle (2011) A virophage at the origin of large DNA transposons. Science 332 (6026) 231-234
- Matthias G. Fischer, Michael J. Allen, William H. Wilson, and Curtis A. Suttle, « Giant virus with a remarkable complement of genes infects marine zooplankton », dans Proceedings of the National Academy of Sciences,vol. 107, no 45, 2010, p. 19508–19513 [lien PMID [archive], lien DOI [archive]]
- http://fr.wikipedia.org/wiki/Virus